Rela
RELA est un gène. Il encode la sous unité p65 de la protéine NF-κB ou nuclear factor kappa B.
Donc RELA = NF-κB-p65
Structure
p65 contient un domaine REL-homology domain (RHD) qui permet la liaison avec l’ADN et un domaine C-terminal transactivation domain (TAD) qui est responsable de l’interaction avec le complexe de transcription basal incluant beaucoup de coactivateurs de transcriptions comme TBP, TFIIB et CREB-CBP.
Phosphorylation
La phosphorylation de p65 joue un rôle dans la régulation de l’activation et la fonction de NF-κB.
p65 peut être phosphorylé soit au niveau du domaine RHD soit le domaine TAD.
Phosphorylation sur le domaine RHD
La phosphorylation du RHD se passe au niveau de la sérine 276 par une protéine kinase A dans le cytoplasme. Elle peut aussi l’être via la MSK1 ou mitogen and stress-activated kinase 1 sous l’induction dans le noyau de TNFα (Tumor necrosis factor alpha). TNFα va retirer la sous unité inhibatrice IκB-a, la permettant d’aller dans le noyau. NF-κB va ensuite être phosphorylée par MSK1. Cela a pour conséquence une réponse augmentée de la transcription des gènes régulés par NF-κB.
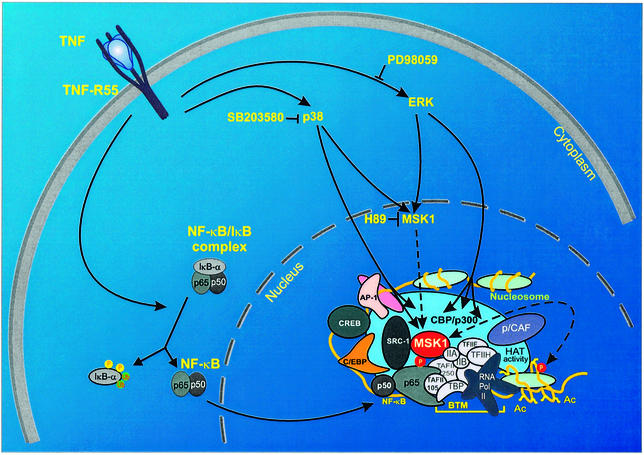
Une phosphorylation du RHD peut aussi se produire sur la sérine 311 par la protéine kinase C.
Phosphorylation sur le domaine TAD
La sérine 529 peut être phosphorylé par une casein kinase II (CKII) après dissociation de la sous unité inhibitrice de NF-κB, IκBα par TNFα.
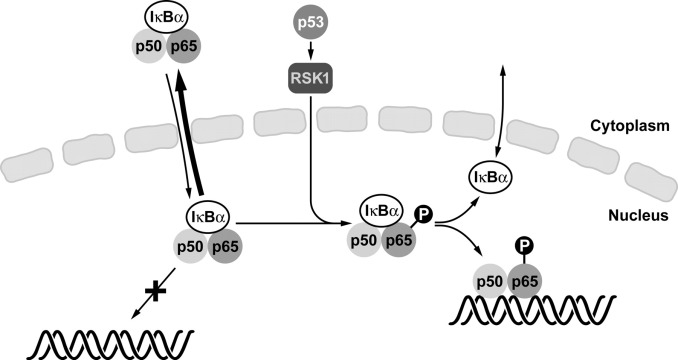
p53 est aussi un régulateur de NF-κB via l’activation de RSK1 qui va entraîner la phosphorylation sur la sérine 536 de p65 et de la partie carboxylterminale. La phosphorylation de la sérine 536 va diminuer son affinité avec IκBα diminuant le niveau d’export nucléaire de la protéine NF-κB.
Acetylation
Des acétylations au niveau de la lysine 221 aide à la dissociation de NF-κB et de IκBα et améliore sa fixation à l’ADN.
L’acétylation de la lysine 310 est essentielle pour une activité transcriptionnelle maximale de p65 sans pour autant impacter sa fixation à l’ADN.
L’acétylation de la lysine 122 et 123 sont, elles, négativement corrélées avec l’activité transcriptionnelle de p65.
Rôles des cibles de p65
- Leukocyte activation & chemotaxis
- Régulation négative du pathway de TNFIKK
- Métabolisme cellulaire
- Processus antigénique
- Inflammation
Système immunitaire
Immunité adaptative et réponse à une invasion de pathogène.
Cancer
Corrélation entre sa localisation nucléaire et le développement de la tumeur.
Inhibiteurs de NF-κB
Bortezomib –> inhibe le protéasome –> Dégradation réduite de IκBα –> inactivation de NF-κB.
NF-κB et LAM
Bortezomib is significantly beneficial for de novo pediatric AML patients with low phosphorylation of the NF-κB subunit RelA
Les patients avec un niveau faible de NF-κB phosphorylé répondent mieux à l’inhibiteur Bortezomib sachant que celui-ci, bloque la dégradation de l’inhibiteur de NF-B.
Les traitements provoquent une demande élevée en NF-κB phosphorylé ?
Nuclear factor-kappaB is constitutively activated in primitive human acute myelogenous leukemia cells
Les LSC in vitro meurent suite à des traitements inhibant NF-κB via un autre inhibiteur du protéasome : proteasome inhibitor MG-132
LAM –> LSC (Cellules souches leucémiques) –> caractérisation –> NF-κB
CD34+ n’expriment pas NF-κB. Les cellules primaires de LAM CD34+ montrent facilement une activité de NF-κB.
- NF-κB actif dans LAM CD34+ / CD38-
- NF-κB basal dans healthy CD34+
Constitutive NF-κB activation in AML: Causes and treatment strategies
Inserm bugged, downloaded using sci-hub
NF-κB activé constitutivement dans les LAMs –> resistance à l’apoptose
Activité du protéosome élevée –> NF-κB plus actif
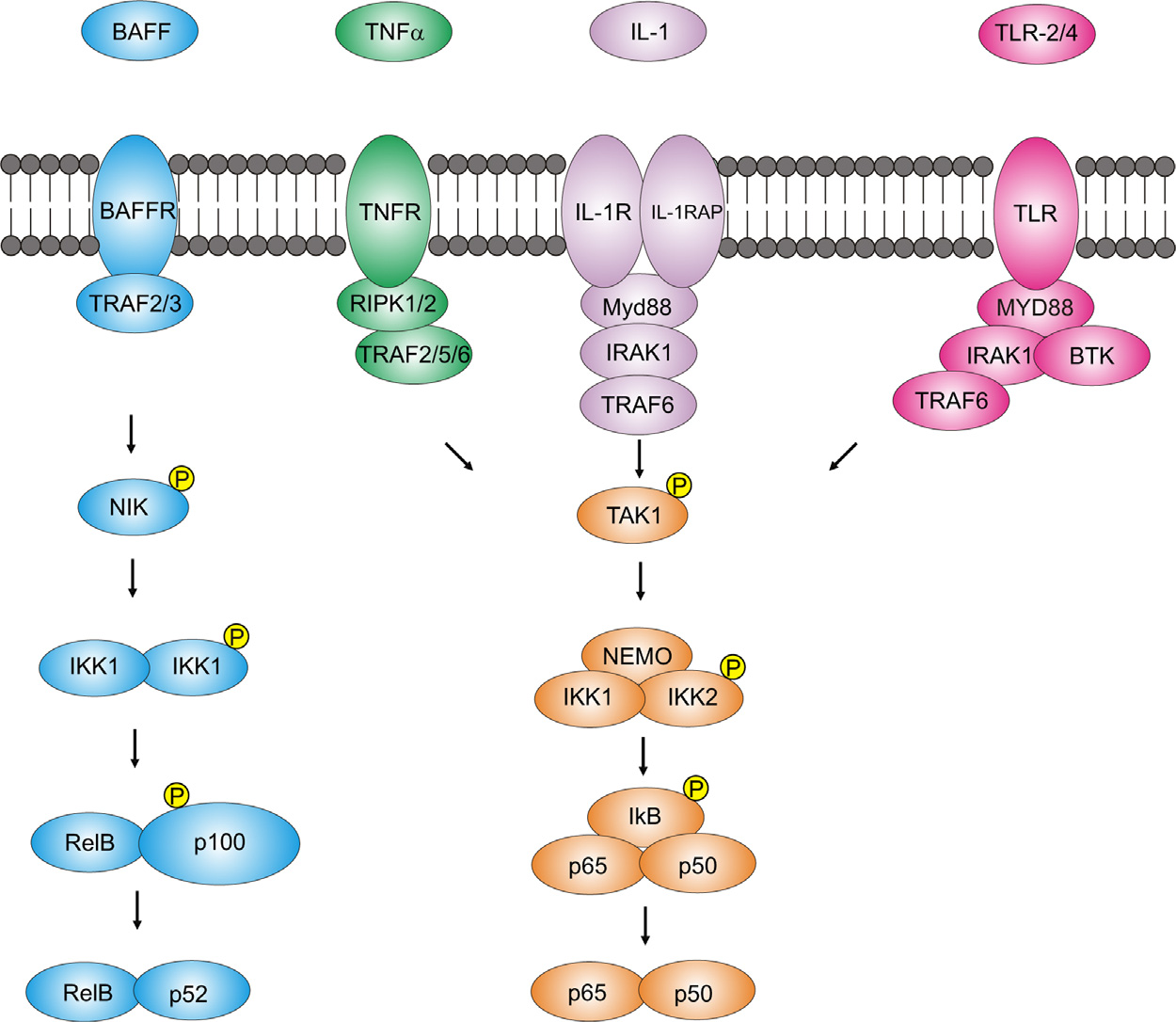
NF-κB signaling
NF-κB activity in normal hematopoiesis
Une activité basal de NF-κB est nécessaire chez les HSCs au niveau du maintien du pull d’HSC mais aussi de leur différenciation en myéloïdes et lymphoïdes.
NF-κB activity in AML
Activité constitutive. Activité la plus élevée dnas les LAM M4 et M5.
- LAM M4 : Myélomonocytaire (Monocyte 20%<x<80%)
- LAM M5 :
Targeted therapy of NF-κB signaling pathways
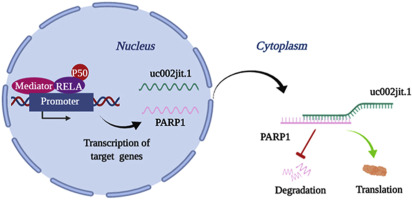
The role of the novel LincRNA uc002jit.1 in NF-kB-mediated DNA damage repair in acute myeloid leukemia cells
Un lncRNA uc002jit.1 est une des targets de RelA. Son expression déstabilise l’ARN de PARP1, diminuant les processus de réparation de l’ADN.